


 微信在线咨询
微信在线咨询

| 产品编号 | 产品名称 | 产品包装 | 产品价格 |
| P2250 | BeyoGold™ GST-tag Purification Resin | 1ml | 65.00元 |
| P2251 | BeyoGold™GST-tag Purification Resin | 10ml | 386.00元 |
| P2253 | BeyoGold™ GST-tag Purification Resin | 100ml | 3076.00元 |
| P2255 | BeyoGold™ GST-tag Purification Resin | 1000ml | 24551.00元 |
碧云天生产的BeyoGold™ GST-tag Purification Resin,即GST(Glutathione S-transferase)标签蛋白纯化介质,俗称GST纯化介质、GST纯化柱或GST柱,是一种用于简单、快速、高效地纯化带有GST标签重组蛋白的纯化介质。
GST,即谷胱甘肽-S-转移酶,与目的蛋白融合表达后,被称为GST标签重组蛋白、GST标签蛋白或GST融合蛋白。GST标签重组蛋白可以通过GST特异性地结合到BeyoGold™ GST-tag Purification Resin,而其它蛋白则不能被结合,含GST标签的重组蛋白可被过量的还原型谷胱甘肽(GSH)溶液洗脱,从而实现GST标签蛋白的分离和纯化。由于纯化过程始终保持在温和的非变性条件下,得到的目的蛋白可以保持其自身的生物学活性,因此可以用于常规的结构和功能研究、抗体制备、以及蛋白与蛋白相互作用、蛋白与核酸相互作用等方面的研究。
本产品不仅可以用于大肠杆菌表达的GST标签重组蛋白的纯化,也可以用于哺乳动物细胞、昆虫细胞及杆状病毒等其它表达系统中表达的GST标签重组蛋白。
GST标签重组蛋白在大肠杆菌中表达时具有可溶性高,并且容易保持目的蛋白完整活性等优点。许多真核蛋白在大肠杆菌中是以包涵体的形式表达的,但当它们在大肠杆菌中与GST融合表达时,相当一部分可以实现可溶性表达,从而有利于后续的纯化。
如果在GST标签和目的蛋白之间含有位点特异性的蛋白酶识别位点,如PreScission Protease, TEV Protease或Thrombin等,则可用相应的蛋白酶切除GST标签。
通常在目的蛋白的N端加上GST标签可以保留GST的酶活性,从而便于使用BeyoGold™ GST-tag Purification Resin进行GST标签重组蛋白的纯化。
本产品采用了一种高度交联的6%琼脂糖凝胶(Beyorose CL-6B,与Sepharose CL-6B性能基本一致)为基质,并使用了进一步优化的接臂和GSH偶联技术,可以高容量、高特异性地结合GST标签重组蛋白。和目前市售的大多数同类产品相比,非特异性蛋白结合更低,耐受压力更强,GSH的偶联更加稳定。
本产品中凝胶的颗粒直径为45-165μm。可耐受的最大压力为0.025MPa,约合5.8psi。采用固定流速进行蛋白纯化时的推荐流速为0.5ml/min。
本产品储存在20%乙醇中,1000ml总体积中500ml为凝胶,500ml为液体。使用时宜把凝胶充分重悬后再吸取。
本产品对含GST标签重组蛋白的结合容量大,其对GST最大结合量为5-6mg蛋白/毫升凝胶,与国际知名品牌的同类产品结合容量相当。实际使用时的最大结合量取决于待纯化的GST标签重组蛋白的分子量大小,分子量越大则最大结合容量越大,分子量越小则最大结合容量越小。对于目的蛋白分子量为50kD的GST标签重组蛋白,每毫升凝胶的实际最大纯化量约为8-12mg,对于目的蛋白分子量为100kD的GST标签重组蛋白,每毫升凝胶的实际最大纯化量约为12-18mg。但对于分子量相同的蛋白,也会因为蛋白本身特性的不同,结合的最大容量也会有所不同。
一个包装的本产品最多可纯化2.5-3.0g GST蛋白。对于目的蛋白分子量为50kD的GST标签重组蛋白,每毫升凝胶的实际最大纯化量约为4.0-6.0g。
包装清单:
| 产品编号 | 产品名称 | 包装 |
| P2255 | BeyoGold™ GST-tag Purification Resin | P2253×10 |
| — | 说明书 | 1份 |
保存条件:
4℃保存,至少一年有效。
注意事项:
请勿在-20℃或更低温度冷冻保存本产品。
保存和纯化过程中应始终保持凝胶湿润。
碧云天有多种不同容量的亲和层析柱空柱管(FCL01/FCL03/FCL06/FCL12/FCL30/FCL60),可以和本产品配合使用。
若离心不能完全除去蛋白样品中的不溶物,可以将样品溶液用0.45μm的滤膜过滤。
GST标签蛋白的纯化应始终保持在非变性条件下,如果融合蛋白以包涵体形式表达,在使用8M尿素或6M盐酸胍溶解包涵体后,需要通过透析去除尿素和盐酸胍并使蛋白复性后才能使用本产品进行纯化。
蛋白样品收集后宜尽快完成纯化工作,并应始终放置在4℃或冰浴,以减缓蛋白降解。为有效抑制蛋白降解,可以在裂解液中添加适量的蛋白酶抑制剂混合物,例如碧云天的P1025/P1026蛋白酶抑制剂混合物(细菌抽提用),或P1005/P1006蛋白酶抑制剂混合物(通用型)。
本产品仅限于专业人员的科学研究用,不得用于临床诊断或治疗,不得用于食品或药品,不得存放于普通住宅内。
为了您的安全和健康,请穿实验服并戴一次性手套操作。
使用说明:
如下以最常见的大肠杆菌中表达纯化GST标签蛋白为例,说明本产品的使用方法。在其它体系中表达时,请参考该表达体系的相关使用说明,并借鉴大肠杆菌中纯化GST标签蛋白的使用说明。
1. 大肠杆菌中可溶性GST标签蛋白的诱导表达
如下以最常用的IPTG诱导表达系统给予说明,诱导表达条件的优化请参照所使用的诱导表达体系的详细说明。其它诱导表达系统请参考适当的使用说明进行。
a. 挑取表达GST标签蛋白的单克隆,接种到3ml或10-20ml含适当抗生素的LB培养液中,37℃培养过夜。
b. 按照1:20的比例取培养过夜的菌液,接种到预热至37℃并含适当抗生素的LB培养液中。例如取5ml培养过夜的菌液接种到100ml预热至37℃并含适当抗生素的LB培养液中。具体的培养体积视需要纯化的蛋白量而定,初步的鉴定培养3-10ml即可;常规的表达纯化,通常可考虑培养100-200ml;制备型的纯化,培养体积可以达到1L或更大。如果希望取得更好的表达效果,建议按照1:100的比例接种过夜培养的菌液,但后续培养至相应的OD值需要更长的时间。
c. 37℃常规培养约30-60min或更长时间,至菌液的OD600达到0.6-1.0。
d. 加入IPTG至终浓度为1mM,37℃诱导表达2-4小时。
注:可以在加入IPTG前取出少量菌液同样培养2-4小时后作为未诱导的对照,也可以在加入IPTG前直接取出少量菌液作为未诱导的对照。对于特定蛋白的诱导表达,最佳的IPTG浓度、诱导温度、和诱导时间需要通过实验确定。
e. 收集菌液至离心管中,4℃ 4,000g离心20分钟或4℃ 15,000g离心1分钟,弃上清,收集沉淀。随后即可进入细菌裂解步骤,也可以在-20℃或-80℃冻存备用。冷冻保存的菌体使用前需置于冰上解冻15分钟。
2. GST标签蛋白的小量纯化
本方法常用于小量样品的快速分析和鉴定,为后续大量制备打下基础。
a. 接步骤1(e),离心收集1ml菌液的细菌沉淀并弃上清,加入100μl裂解缓冲液,将细菌沉淀充分重悬于裂解缓冲液中,可进行轻微的vortex(尽量避免产生气泡)。
注:根据GST标签蛋白表达的丰度,菌液和裂解缓冲液的体积比可以在25:1-5:1范围内适当调整。表达丰度非常高时,每毫升菌液沉淀可以加入200μl裂解缓冲液;表达丰度非常低时,每毫升菌液沉淀可以加入40μl裂解缓冲液。相关溶液的配制方法附后。如有必要,在裂解细菌之前,可以在裂解液中添加适量的蛋白酶抑制剂混合物,例如碧云天的P1025/P1026蛋白酶抑制剂混合物(细菌抽提用),或P1005/P1006蛋白酶抑制剂混合物(通用型)。
b. 加入溶菌酶至1mg/ml并轻轻混匀,尽量避免产生气泡,冰水浴或冰上放置30min。
注:如果没有溶菌酶,也可以直接冰上超声裂解细菌,随后进入步骤2d。超声功率200-300W,每次超声处理10s,每次间隔10s,共超声处理6次。具体超声处理的方式须根据特定型号的超声仪器自行摸索和优化。
c. 轻轻vortex数下,以充分裂解细菌,尽量避免产生气泡。
d. 4℃离心(15000g×10min),取10μl上清留样作后续检测用,收集余下上清至一新的洁净离心管中。
注:本步骤及后续步骤收集的上清必须保持澄清,即不含任何不溶物。上清中如果混有不溶性杂质会严重影响后续纯化获得蛋白的纯度。
e. 加入20μl混合均匀的50% BeyoGold™ GST-tag Purification Resin,4℃在摇床上缓慢摇动30min,以充分结合带GST标签的目的蛋白。
注:缓慢摇动30min已经可以确保蛋白充分结合,但可以根据时间安排的需要缓慢摇动更长时间甚至缓慢摇动过夜。经测试,直接使用50% BeyoGold™ GST-tag Purification Resin也能获得良好的纯化效果。但如果希望获得更高的标签蛋白得率,可以参考步骤3f,用与凝胶等体积的裂解缓冲液平衡BeyoGold™ GST-tag Purification Resin 2-3次。平衡后,待纯化蛋白的得率通常会有所提高。
f. 4℃离心(1000g×10s)沉淀凝胶,取20μl上清留样作后续检测用,其余上清弃去。
g. 加入100μl 裂解缓冲液重悬凝胶,4℃离心(1000g×10s),取20μl上清留样作后续检测用,其余上清弃去。
h. 重复步骤g,再进行一次洗涤。
注:如有需要,可以根据具体情况使用PreScission Protease (P2302/P2303)、TEV Protease (P2307/P2308)或Thrombin等酶切融合蛋白的GST标签从而释放目的蛋白。详细的GST标签酶切操作请参考步骤6,GST标签的酶切。
i. 加入20μl洗脱缓冲液,轻轻重悬凝胶。4℃离心(1000g×10s),收集上清及凝胶。上清即为纯化获得的带有GST标签的目的蛋白。
j. 重复步骤i两次。共洗脱并收集约60μl纯化的蛋白样品。
3. GST标签蛋白的大量纯化
本方法适用于较大量(例如菌液体积在100ml及以上)蛋白样品的纯化。
a. 接步骤1(e),对于新鲜的或解冻的细菌沉淀,按照每克细菌沉淀湿重加入2-5ml的比例加入裂解缓冲液,充分重悬菌体。如有必要,可以在裂解细菌之前,在裂解液中添加适量的蛋白酶抑制剂混合物,例如碧云天的P1025/P1026蛋白酶抑制剂混合物(细菌抽提用),或P1005/P1006蛋白酶抑制剂混合物(通用型)。
b. 加入溶菌酶至终浓度为1mg/ml并混匀,冰水浴或冰上放置30min。
注:如果没有溶菌酶,也可以直接进入步骤3c。
c. 冰上超声裂解细菌。超声功率200-300W,每次超声处理10s,每次间隔10s,共超声处理6次。
注:具体超声处理的方式须根据特定型号的超声仪器自行摸索和优化。
d. (可选做)如果超声处理后裂解液非常粘稠,可以加入RNase A至10μg/ml及DNase I至5μg/ml,冰上放置10-15min。或者也可以使用适当的装好了较细针头的注射器,反复抽吸数次,以剪切粘稠的基因组DNA等。
e. 4℃ 10,000g离心20-30min,收集细菌裂解液上清并置于冰水浴或冰上。可以取20μl上清留作后续检测用。
注:上清必须保持澄清,即不含任何不溶物,才能进行下一步的纯化。上清中如果混有不溶性杂质会严重影响后续纯化获得蛋白的纯度。
f. 取适量混合均匀的BeyoGold™ GST-tag Purification Resin,4℃离心(1000g×10s)弃去储存液,向凝胶中加入等体积的裂解缓冲液以重悬并平衡凝胶,4℃离心(1000g×10s)弃去液体,再重复平衡1-2次,弃去液体。按照每0.5ml凝胶(相当于1ml 50%的凝胶)中加入4ml细菌裂解液上清的比例(1:8),混合细菌裂解液上清和BeyoGold™ GST-tag Purification Resin。4℃在侧摆摇床或水平摇床上缓慢摇动60min。
g. 将裂解液和BeyoGold™ GST-tag Purification Resin的混合物装入适当的空柱管中,如碧云天的亲和层析柱空柱管。
注:也可先装柱,用1倍柱体积的裂解缓冲液平衡后加入细菌裂解液上清,后续可以把流穿液收集后重复上柱3-5次以充分结合目的蛋白。先混合后装柱的方式操作起来相对麻烦一些,但更有利于GST标签蛋白与填料的充分结合。
h. 将纯化柱底部的盖子打开,在重力作用下使柱内液体流出,收集约20微升流穿液作后续分析用。
i. 洗柱5次,每次加入1-2个柱体积的裂解缓冲液,每次均收集约20微升穿柱的液体用于后续的分析检测用。例如1ml混合均匀的50% BeyoGold™ GST-tag Purification Resin装柱后的柱体积为0.5ml,即1ml混合均匀的50% BeyoGold™ GST-tag
Purification Resin装柱后每次洗柱的裂解缓冲液体积为0.5ml。洗柱及下一步洗脱过程中可以用Bradford法(P0006)简单快速地检测每次洗涤液和洗脱液中的蛋白含量,从而考虑增加或减少洗涤和洗脱的次数。
注:如果出现后续获得蛋白纯度不够高的情况,可以再增加洗柱次数2-3次。若有需要,也可以用PreScission Protease (P2302/P2303),TEV Protease (P2307/P2308)或Thrombin等酶切融合蛋白的GST标签从而释放目的蛋白。详细的GST标签酶切操作请参考步骤6,GST标签的酶切去除。
j. 洗脱目的蛋白6-10次,每次用一个柱体积的洗脱缓冲液。将每次的洗脱液分别收集到不同的离心管中。收集获得的洗脱液即为纯化的GST标签蛋白样品。蛋白纯化效果可以参考图1。
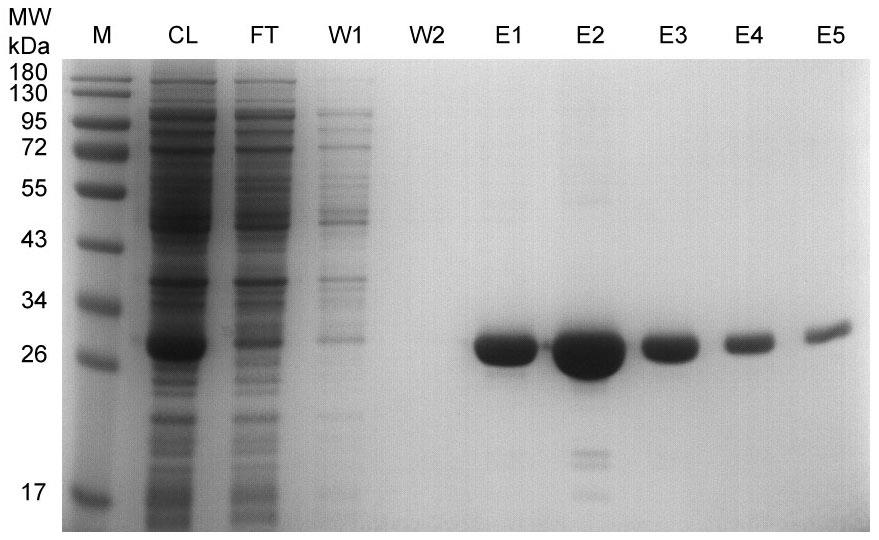
图1. GST标签蛋白纯化效果图。M:marker;CL:细菌裂解液(cell lysate);FT:上样流穿液(flow through);W1-W2:洗涤液1-2 (wash 1-2); E1-E5:洗脱液1-5 (elution 1-5)。注:实际的电泳结果会因样品、上样量等的不同而有所不同。
4. FPLC(Fast Protein Liquid Chromatography)纯化
a. 将所有材料平衡至纯化操作所需的温度。
b. 用裂解缓冲液冲洗纯化柱的底部,除去空气。确保纯化柱滤网下没有任何空气。
c. 关闭纯化柱的出水口,柱内保留1-2cm高度的裂解缓冲液。
d. 轻轻晃动凝胶使其充分重悬,用移液器吸取适当体积的凝胶并转移至纯化柱内,转移凝胶时将悬液沿纯化柱内壁小心倒入以减少气泡的产生。
e. 立即用裂解缓冲液灌满纯化柱,上部接好顶部装置,并将纯化柱与泵相连接。
f. 打开纯化柱的出水口,开启泵并以所需流速运行。当柱床体积不再变化后,保持装柱的流速至少再平衡3个柱床体积,并在纯化柱上标记柱床的高度。
注:纯化过程中,流速不宜超过装柱流速的75%。理想情况下,装柱的BeyoGold™ GST-tag Purification Resin的柱压应不超过1bar(0.1MPa)。
g. 关闭泵及纯化柱的出水口,卸下顶部装置,小心的将纯化柱内灌满裂解缓冲液。
h. 将适配器以一定角度插入纯化柱柱床的上方,确保它们之间没有气泡。
i. 将适配器慢慢下移(保持适配器出水口打开),直到标记的位置(步骤g),将适配器锁定在此位置。将纯化柱与泵或层析系统连接,开始柱平衡。若有需要,可以调整适配器的位置。
j. 用大约5-10倍柱体积的裂解缓冲液平衡纯化柱。
k. 将离心过滤后澄清的细菌裂解液通过FPLC系统的泵以适当流速通过纯化柱。
注:流速是影响GST标签蛋白与BeyoGold™ GST-tag Purification Resin结合的重要因素之一。因为GST与GSH结合较慢,当细菌裂解液过柱时,保持较慢的流速对于蛋白与BeyoGold™ GST-tag Purification Resin的最大结合十分重要,推荐使用的流速为0.5-1ml/min。蛋白的性质、pH及温度也会影响结合能力。细菌的裂解方法可参考步骤3中的相关部分。
l. 用5-10倍柱体积的裂解缓冲液洗柱,或直到洗涤后的液体中检测不到蛋白成分,取少量洗涤后的液体留样。
m. 用5-10倍柱体积的洗脱缓冲液洗脱。
注:每次纯化所需的洗脱液体积都有可能不同,有时可能需要用高浓度的谷胱甘肽进行额外的洗脱。纯化过程中的流穿液,洗涤液和洗脱液应该收集以备SDS-PAGE和Western Blot检测。
5. GST pull-down
本产品可以用于蛋白-蛋白相互作用的研究。通过GST标签固定在BeyoGold™ GST-tag Purificatioin Resin上的蛋白作为“诱饵”,可以捕获能够与其相互作用的靶蛋白,即“猎物”。GST pull-down实验大致有两种用途,其一是鉴定已知GST标签蛋白与另一蛋白的相互作用;其二是大量捕获各种可能的与“诱饵”蛋白有相互作用的多种蛋白,以用于质谱分析、检测和鉴定。
a. 轻轻摇动BeyoGold™ GST-tag Purification Resin,使其完全重悬,吸取适量50%的凝胶悬液至离心管中(每个样品需要50μl 50%的凝胶悬液),加入20倍体积的GST pull-down蛋白结合缓冲液(例如50μl 50%的凝胶悬液加入1ml蛋白结合缓冲液),颠倒混匀以平衡凝胶。1000g离心2分钟,小心吸除管内液体。加入与凝胶等体积的GST pull-down蛋白结合缓冲液重悬凝胶以获得平衡好的50%凝胶悬液(例如起始为50μl 50%的凝胶悬液加入25μl GST pull-down蛋白结合缓冲液重悬)。
b. 参考下表,依次加入GST pull-down蛋白结合缓冲液、经步骤a平衡好的BeyoGold™ GST-tag Purification Resin、靶蛋白和GST标签蛋白(诱饵蛋白)或GST蛋白(阴性对照)。
| 组份 | 阴性对照1 | 阴性对照2 | 样品管 |
| GST pull-down蛋白结合缓冲液 | 400μl | 400μl | 400μl |
| 平衡好的50%凝胶悬液 | 50μl | 50μl | 50μl |
| 靶蛋白样品 | 25μg | 25μg | 25μg |
| GST蛋白(对照) | - | 25μg | - |
| GST标签蛋白(诱饵蛋白) | - | - | 25μg |
| GST pull-down蛋白结合缓冲液 | 补足至相同的终体积(0.5-1ml) | ||
c. 颠倒混匀各管,置于侧摆摇床上,4℃孵育2h或过夜。
d. 4℃ 1000g离心2分钟,将上清转移至新的离心管中,留作后续分析。
e. 加入100μl GST pull-down蛋白结合缓冲液重悬凝胶,4℃离心(1000g×10s),小心吸除上清。
f. 重复步骤e 2次,以充分洗去未结合的蛋白。
g. 加入50μl GST pull-down洗脱缓冲液,轻轻晃动并孵育10分钟。
注:也可以直接向凝胶中加入25μl SDS-PAGE上样缓冲液(2X),煮沸5分钟后离心(12000g×1min)取上清用于后续检测。
h. 4℃ 1000g离心2分钟,小心吸取上清用于后续检测。后续常用于SDS-PAGE后考马斯亮蓝染色、银染或者Western检测。
6. GST标签的酶切去除
大多数情况下,融合表达的GST标签不会影响目的蛋白的活性,可以用完整的融合蛋白进行活性检测。如果GST标签可能影响目的蛋白的活性,那么在构建质粒时可以在GST标签和目的蛋白之间加入适当的蛋白酶的识别位点,例如PreScission Protease、TEV Protease或Thrombin等的识别位点,这样就可以在柱上(on column)或洗脱后用相应的蛋白酶切除GST标签。由于PreScission Protease能在4℃进行高效的酶切,而TEV Protease或Thrombin的最佳酶切温度是室温而且耗时较长,因此我们推荐在设计目的蛋白的GST标签时,添加PreScission Protease识别的酶切位点。具体关于如何添加PreScission Protease识别的酶切位点,可以参考PreScission Protease (P2302/P2303)的产品说明书。PreScission Protease (P2302/P2303)可以从碧云天购买。PreScission Protease(带有GST标签)酶切后仅不含GST标签的目的蛋白被洗脱,PreScission Protease、GST标签都会留在凝胶柱上。如下就如何使用PreScission Protease进行GST标签的切除进行详细的说明。
a. 柱上酶切含GST标签的融合蛋白(以8mg GST标签蛋白/ml凝胶为例)
(a) 在GST标签蛋白结合于纯化柱(1ml)并用洗涤液充分洗涤后,再用10倍柱床体积PreScission Protease的酶切缓冲液平衡柱子。PreScission Protease的酶切缓冲液的组分为50mM Tris-HCl,150mM NaCl,1mM EDTA,1mM DTT, pH7.5。
(b) 准备PreScission Protease:约每毫克GST标签蛋白使用20U PreScission Protease。对于8mg GST标签蛋白需使用160U PreScission Protease,用PreScission酶切缓冲液稀释至与凝胶柱相同的体积,即1ml。
(c) 将稀释好的1ml PreScission Protease泵入纯化柱中,4℃保持4-8h(为确保酶切完全,可以4℃酶切过夜)。如果蛋白结合是在离心管中进行的,可将准备好的PreScission Protease直接加入离心管中,4℃在摇床上缓慢摇动4-8小时(为确保酶切完全,可以4℃酶切过夜)。
(d) 用1倍柱床体积的PreScission Protease酶切缓冲液洗涤,重复三次,分别收集每次的洗涤液。如果酶切反应是在离心管中进行的,1000g离心2分钟,收集上清,然后加入1ml酶切缓冲液重悬沉淀,离心(1000g×2min)收集上清,接着再加入1ml酶切缓冲液重悬沉淀,离心(1000g×2min)收集上清。洗脱组分中含有切除了GST标签的目的蛋白,而GST标签和带有GST标签的PreScission Protease则仍然结合在凝胶柱上。
b. 洗脱后酶切含GST标签的融合蛋白(以8mg GST标签蛋白/ml凝胶为例)
(a) 使用脱盐柱快速除去洗脱组分中的GSH,或用PreScission Protease酶切缓冲液进行透析。
(b) 按每100μg GST标签蛋白加入2U PreScission Protease的比例加入蛋白酶,如果蛋白未定量,可以按照每1ml凝胶加入160U PreScission Protease (按照每毫升凝胶结合8mg GST标签蛋白进行预估)的比例进行。4℃孵育4h。
(c) 将酶切后的蛋白样品加入预先用PreScission Protease酶切缓冲液平衡好的BeyoGold™ GST-tag Purification Resin,室温结合20-30分钟。
(d) 500g离心5分钟,收集上清,其中含有切除了GST标签的目的蛋白,没有消化的GST标签蛋白和PreScission Protease则结合在凝胶沉淀中。
7. BeyoGold™ GST-tag Purification Resin的再生
当BeyoGold™ GST-tag Purification Resin的蛋白结合能力下降时,其原因可能是由于蛋白沉淀、变性蛋白或非特异结合蛋白积累在介质上导致,此时可通过以下方法再生。
a. 去除蛋白沉淀或变性蛋白:
(a) 用2倍柱体积的6M盐酸胍洗柱。
(b) 随后立即用5倍柱体积的PBS(pH 7.3)洗柱。
b. 去除疏水结合的蛋白:
(a) 用3-4倍柱体积的70%乙醇或2倍柱床体积的1% Triton X-100洗柱。
(b) 随后立即用5倍柱体积的PBS (pH 7.3)洗柱。
(c) 再生后的凝胶,用20%乙醇平衡后,4℃保存。
常见问题:
1. 裂解液中目的蛋白含量过低。
a. 目的蛋白表达在包涵体中。
需调整表达条件(如降低蛋白表达的温度和使用较低浓度的IPTG进行蛋白的诱导),使目的蛋白至少部分实现可溶性表达或使用变性剂溶解包涵体后复性,然后再进行纯化。
b. 培养条件不正确。
在同样的蛋白表达条件下,使用目的蛋白克隆时使用的含GST标签的空载体(例如碧云天的D2913 pET-N-GST-PreScission、D2912 pET-N-GST-Thrombin-C-His、D2926 pT7Duet-N-GST或D2927 pT7Duet-N-GST-PreScission)进行GST表达,观察其表达是否正常。
c. 诱导表达的蛋白迅速降解。
设置不同的诱导表达时间,摸索细菌培养、蛋白表达和降解的时间,最终选择适当的诱导时间。若蛋白是在细菌裂解后降解,则需加入蛋白酶抑制剂。另外,在蛋白纯化的所有步骤中,须严格保持在4℃左右,以尽量减缓蛋白降解。
2. 表达的GST标签蛋白蛋白不与BeyoGold™ GST-tag Purification Resin结合。
a. 超声导致融合蛋白变性。
超声强度过高会导致融合蛋白变性,从而不能很好地与BeyoGold™ GST-tag Purification Resin结合。此时宜使用温和的超声条件,并且超声过程必须在冰浴中进行。另外也可以考虑采用非超声的裂解条件。
b. 目的蛋白由于还原性不足,导致空间构象异常。
在裂解前向裂解缓冲液中加入DTT至终浓度1-10mM,这样能显著改善某些蛋白的空间结构,从而有效改善某些GST标签蛋白与GST-tag Purification Resin的结合。
c. 检测空载体表达的GST与BeyoGold™ GST-tag Purification Resin的结合能力,以确定纯化体系能否正常工作。
表达GST的细菌裂解后,用BeyoGold™ GST-tag Purification Resin纯化其中的GST。如果空载体表达的GST能被BeyoGold™ GST-tag Purification Resin很好地纯化,说明纯化体系是可以正常工作的。融合蛋白可能改变了GST的构象或遮蔽了GST,从而抑制了其与BeyoGold™ GST-tag Purification Resin的结合。可以尝试将蛋白结合温度降低至4℃并减少洗柱的次数来改善,有些蛋白同2b中提到的加入适量的DTT也有可能改善纯化效果。
d. 使用平衡好的BeyoGold™ GST-tag Purification Resin用于GST标签蛋白的纯化。
GST标签蛋白与BeyoGold™ GST-tag Purification Resin的结合在pH6.5以下或pH8.0以上会显著下降。在将蛋白裂解液与BeyoGold™ GST-tag Purification Resin结合前,请用裂解缓冲液充分平衡。
e. 使用未使用过的BeyoGold™ GST-tag Purification Resin.
如果BeyoGold™ GST-tag Purification Resin已被多次使用,其与GST标签蛋白的结合能力可能有所下降。在发现GST标签蛋白不能有效结合的情况下,有必要使用未使用过的BeyoGold™ GST-tag Purification Resin,或将BeyoGold™ GST-tag
Purification Resin再生后再尝试使用。
f. 降低蛋白样品上柱时的流速。
影响GST标签蛋白与与纯化介质结合的一个非常重要的参数是流速,GST标签蛋白与GSH的结合速度相对较慢,因此在样品上柱时保持较慢的流速对于GST标签蛋白的充分结合非常重要。
3. GST标签蛋白不能被有效洗脱。
a. 使用新鲜配制的洗脱缓冲液。
b. 增加洗脱时间以改善洗脱效果。对于纯化柱的洗脱可以降低洗脱时的流速。
c. 增加洗脱缓冲液的用量。有时,尤其是在柱上通过酶切去除GST标签的情况,需要使用更大量的洗脱缓冲液进行洗脱。
d. 增加洗脱缓冲液中GSH的浓度。
洗脱缓冲液中含有的10mM GSH在大多数情况下是足够进行洗脱的,但也存在洗脱不完全的特殊情况。对于这种情况,可以将洗脱缓冲液中的GSH浓度提高至20-50mM。
e. 提高洗脱缓冲液的pH。
较低的pH会影响洗脱效率。在洗脱效率较低时,可以尝试提高洗脱缓冲液的pH至8-9。
f. 增加洗脱缓冲液中的离子强度。
离子间相互作用可能会将蛋白留在柱上,增加洗脱缓冲液中的离子强度会改善这种状况。通常可以向洗脱缓冲液中加入0.1-0.2M NaCl。
g. 向洗脱缓冲液中加入非离子型去垢剂。
非特异的疏水作用可能会使蛋白沉淀,并阻止目的蛋白的洗脱。在此情况下,加入适当的去垢剂可能会有所改善。向洗脱缓冲液中加入Triton X-100至终浓度为0.1%或N-octylglucoside至终浓度为2%,都能够显著改善某些GST标签蛋白的洗脱。
4. 纯化获得的GST标签蛋白经SDS-PAGE电泳/Western blot检测有多条条带。
a. 洗涤条件错误或洗涤不充分。检查纯化过程中是否严格按照说明书的建议进行了充分的的洗涤。
b. 70kD蛋白与GST标签蛋白共纯化。
70kD蛋白可能是大肠杆菌dnaK基因的表达产物,该蛋白参与大肠杆菌的蛋白折叠过程。据报道它们之间的这种结合可以通过在上柱前,将GST标签蛋白在含有ATP的缓冲液(50mM Tris-HCl, 2mM ATP, 10mM MgSO4, pH7.4)中37℃孵育10分钟而得以消除。也可以在洗涤时用上述含有ATP的缓冲液洗涤去除,或者后续通过离子交换柱等方法去除70kD蛋白。
c. 加入蛋白酶抑制剂。
多条条带有可能是由于GST标签蛋白的部分降解产生的。向裂解液中加入适当的蛋白酶抑制剂有时会有所改善。
注:在使用Thrombin或factor Xa酶切GST标签蛋白时,溶液中的丝氨酸蛋白酶抑制剂前必须除去,否则会严重抑制酶切效果。Prescission Protease通常不被认为是典型的丝氨酸蛋白酶,并且对多种蛋白酶抑制剂不敏感。
d. 使用蛋白酶缺陷型菌株。
多条条带的出现可能是宿主细菌表达的蛋白酶对目的蛋白的酶切结果。如果是这种情况,需要使用蛋白酶缺陷型菌株(如BL21(ompT))进行表达。
e. 降低超声强度。
细胞破碎效果可以通过溶液的澄清度或显微镜下观察细菌形态来判断。超声前加入溶菌酶通常可以改善超声破碎效果。尽量注意避免超声过程中产生泡沫,因为这可能会导致GST标签蛋白的变性。过度超声可能会导致宿主蛋白的变性及与GST标签蛋白的共纯化。
f. 采用额外的纯化步骤。
非特异条带可能是与GST标签蛋白与各种分子伴侣的共纯化,因为它们参与大肠杆菌中新合成蛋白的正确折叠。这些蛋白包括但不限于DnaK(70kD)、DnaJ(37kD)、GrpE(40kD)、GroEL(57kD)和GroES(10kD)。这种情况下,可以考虑采用额外的其它方法进一步纯化目的蛋白。
g. 抗GST的抗体中检测GST标签蛋白出现很多杂带时可能识别大肠杆菌的某些蛋白。
所使用的抗GST抗体可能能够与大肠杆菌某些蛋白结合,从而导致Western检测时出现杂带,选择经过大肠杆菌蛋白吸附过的抗GST抗体,就能很好地减少甚至消除这些杂带。
5. GST标签蛋白不能被完全酶切。
a. PreScission Protease,TEV Protease或Thrombin等酶的用量不正确。
检查酶切体系中GST标签蛋白的量,从而确定蛋白酶的用量。通常合适的用量为,PreScission Protease至少10U/mg GST标签蛋白,TEV Protease至少300U/mg GST标签蛋白,Thrombin至少10U/mg GST标签蛋白,Factor Xa,酶与GST标签蛋白的比率至少是1%(w/w)。在某些情形下,酶切时使用1mg/ml的GST标签蛋白可以达到比较理想的酶切效果。向反应缓冲液中加入少量SDS(不超过0.5%,w/v)能够显著提高factor Xa对某些GST标签蛋白的酶切效果。
b. 增加酶切时间和提高酶的浓度。
如果GST标签蛋白没有被完全酶切,可以尝试延长酶切时间至20小时或更久。酶量也可酌情增加。
c. 确认蛋白酶特异识别的位点存在。
检查设计构建的DNA序列,并与测序结果相比对,确认所使用酶的特异识别位点是正确的。
d. 确保体系中没有相应蛋白酶的抑制剂。
酶切前,对于在凝胶上的酶切可以用相应的酶切缓冲液平衡,对于在溶液中的酶切可以先用酶切缓冲液充分透析或者脱盐后更换成相应的酶切缓冲液。
6. 酶切后的目的蛋白电泳分析发现多条条带。
a. 确定额外条带是何时出现的。
如果检测确认酶切前这些条带是不存在的,那么这些条带就是酶切后新产生的,这很有可能是GST标签蛋白在宿主细胞中发生降解导致的。如果酶切前这些条带就存在,参考第4点中提到的解决办法。
b. 目的蛋白可能含有PreScission Protease,TEV Protease或Thrombin等的识别位点。此时需要仔细分析基因序列,如果确实存在,就需要考虑重新构建质粒,更换使用不同的蛋白酶。
7. GST pull-down过程中的问题。
a. 信号偏弱。
(a) 可能样品中作为猎物的靶蛋白含量不足,如有可能,请检测样品中是否含有足够量的靶蛋白。
(b) 如果样品中靶蛋白丰度没有问题,则有可能是蛋白结合条件不利于靶蛋白与GST标签蛋白的结合,改变结合溶液中的盐及去垢剂的种类和浓度可能会有所帮助。
(c) 如果是洗脱效率低,那么蛋白复合体可能还留在BeyoGold™ GST-tag Purification Resin上,可将其与纯化柱等体积的SDS-PAGE上样缓冲液(2X)共同煮沸后取上清进行后续检测。如果是此原因导致的信号偏低,可以通过增加洗脱时间、提高洗脱缓冲液中GSH浓度(20-50mM)来改善。
b. 有非特异性的背景。
样品中的蛋白可能与GST或BeyoGold™ GST-tag Purification Resin产生非特异结合。和凝胶结合时,减少裂解液的上样量,增加洗涤液的用量并增加洗涤次数,通常对能在一定程度上降低非特异性的背景。另外,适当提高洗涤液中的离子强度或添加适量的适当去垢剂,也会对降低背景有所帮助。
附录:
相关溶液的配制方法:
| a. | 裂解缓冲液: | d. | GST pull-down蛋白结合缓冲液* | ||
| PBS | Tris-HCl | 50mM | |||
| NaCl | 140mM | NaCl | 200mM** | ||
| KCl | 2.7mM | EDTA | 1mM** | ||
| Na2HPO4 | 10mM | NP-40 | 1%(v/v)** | ||
| KH2PO4 | 1.8mM | DTT | 1mM** | ||
| pH 7.3 | MgCl2 | 10mM** | |||
| b. | 洗脱缓冲液: | pH 8.0 | |||
| Tris-HCl | 50mM | e. | GST pull-down洗脱缓冲液* | ||
| GSH | 10mM | Tris-HCl | 50mM | ||
| pH 8.0 | NaCl | 400mM** | |||
| c. | PreScission酶切缓冲液 | GSH | 50mM | ||
| Tris-HCl | 50mM | EDTA | 1mM** | ||
| NaCl | 150mM | DTT | 1mM** | ||
| EDTA | 1mM | pH 8.0 | |||
| DTT | 1mM | *可添加适量的蛋白酶抑制剂,例如碧云天的P1025/P1026或P1005/P1006。 | |||
| pH 7.5 | |||||
| **可以根据具体的实验要求及蛋白作相应的改变。 | |||||
相关产品:
| 产品编号 | 产品名称 | 包装 |
| P2210 | BeyoGold™ His-tag Purification Resin | 10ml |
| P2218 | BeyoGold™ His-tag Purification Resin | 100ml |
| P2220 | BeyoGold™ His-tag Purification Resin | 1000ml |
| P2226 | His标签蛋白纯化试剂盒 | 10ml |
| P2251 | BeyoGold™ GST-tag Purification Resin | 10ml |
| P2253 | BeyoGold™ GST-tag Purification Resin | 100ml |
| P2255 | BeyoGold™ GST-tag Purification Resin | 1000ml |
| P2262 | GST标签蛋白纯化试剂盒 | 10ml |
| P1005 | 蛋白酶抑制剂混合物(通用型) | 1ml |
| P1006 | 蛋白酶抑制剂混合物(通用型) | 5ml |
| P1025 | 蛋白酶抑制剂混合物(细菌抽提用) | 1ml |
| P1026 | 蛋白酶抑制剂混合物(细菌抽提用) | 5ml |
| P1030 | 蛋白酶抑制剂混合物(His-Tag蛋白纯化用) | 1ml |
| P1031 | 蛋白酶抑制剂混合物(His-Tag蛋白纯化用) | 5ml |
| P2302 | PreScission Protease | 100U |
| P2303 | PreScission Protease | 500U |
| D2911 | pET-N-GST-Thrombin-C-His | 1μg |
| D2916 | pET-N-GST-PreScission | 1μg |
| D2931 | pET-Dual-N-GST | 1μg |
| D2933 | pET-Dual-N-GST-PreScission | 1μg |
| AG768 | GST抗体 | >20次 |
| AF0174 | GST Mouse Monoclonal Antibody | 50μl |









 微信在线咨询
微信在线咨询












